Оглавление:
- Автор John Day day@howwhatproduce.com.
- Public 2024-01-30 11:52.
- Последнее изменение 2025-01-23 15:05.
Авторы: Мадхумита Каннан, Генри Нгуен, Эшли Уррутия Авила, Мей Джин
Этот код MATLAB представляет собой программу для определения точного размера, формы и местоположения опухоли, обнаруженных на снимках МРТ головного мозга пациента. Эта программа изначально предназначена для работы с обнаружением опухолей при МРТ головного мозга, но ее также можно использовать для диагностики рака при сканировании других органов.
В следующих инструкциях сначала будут описаны методы анализа изображений путем фильтрации и очистки МРТ-сканирования, преобразования в двоичную форму, медианной фильтрации и скользящих окон. Затем он проинструктирует, как изолировать опухоль, используя предварительно созданную эллиптическую маску, и ее дальнейшую фильтрацию, чтобы очертить периметр формы опухоли.
После обнаружения опухоли инструкции будут дополнительно описывать, как включить эту программу в графический интерфейс пользователя (GUI). К этим инструкциям будут приложены соответствующий код и файлы, которые помогут объяснить, как работает этот анализ МРТ.
Некоторые вещи, которые следует знать, скачать и подготовить, прежде чем приступить к выполнению этой инструкции: 1. Убедитесь, что у вас загружена последняя версия MATLAB. Вы можете установить R2018b здесь:
2. Чтобы запустить эту программу, вам необходимо иметь доступ к файлам МРТ-сканирования мозга. Хотя некоторые из них всегда можно найти на изображениях Google, тщательный и точный анализ может быть выполнен на основе соответствующих изображений различных слоев сканированных изображений мозга каждого пациента. Вы можете получить доступ к файлам для 20 различных пациентов с глиобластомой до и после лечения из этой базы данных:
3. Фокус этой программы и различные методы, которыми руководствуется этот проект, изложены в этом исследовательском документе:
Шаг 1. Запустите графический интерфейс пользователя (GUI)
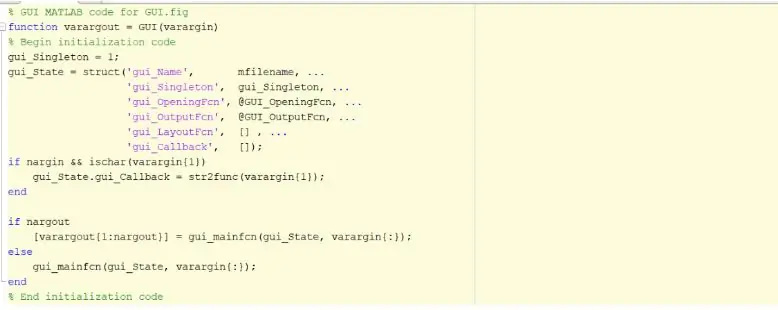
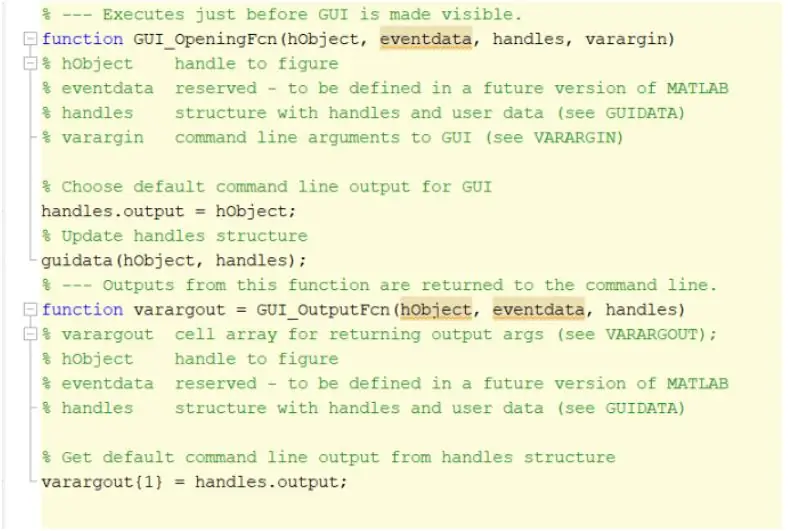
Первым шагом будет создание и запуск графического пользовательского интерфейса, GUI. Это можно сделать, введя руководство в командное окно, нажав клавишу ВВОД и создав новый графический интерфейс. После завершения этого шага вы можете начать создавать такие функции, как оси, статический текст, редактировать текст и кнопки, которые будут отображаться после запуска программы, и пользователь сможет взаимодействовать с ней. Эти функции можно редактировать и управлять ими с помощью инспектора свойств, однако наиболее важной особенностью, которую необходимо изменить при создании этих функций, является имя тега. Важно изменить имя тега каждой реализованной функции, потому что это позволит нам создать различимую функцию обратного вызова. Как только вы будете удовлетворены макетом вашего графического интерфейса, вы можете перейти к загрузке файлов DICOM, которые будут отображаться в графическом интерфейсе.
Шаг 2: Загрузка и чтение изображений МРТ в MATLAB
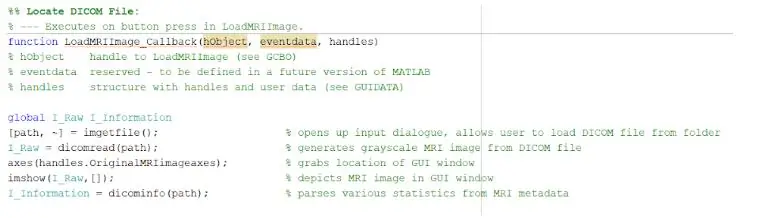
Чтобы загрузить файлы DICOM, вам необходимо правильно инициализировать функцию обратного вызова, которая будет выполняться при нажатии кнопки «Загрузить изображение МРТ». Как только это будет завершено, вы должны создать глобальную переменную, которая будет отображать изображение на осях ручек, где вы хотите, чтобы отображалось исходное изображение МРТ. Все изображения сканирования МРТ, загруженные из базы данных, представляют собой файлы в формате DICOM, которые необходимо загрузить в каталог MATLAB. Найдите файл с помощью imgetfile, чтобы загрузить их в программу. Изображения считываются с использованием встроенной функции MATLAB «dicomread», и первое необработанное изображение для каждого файла вставляется в левые оси графического интерфейса пользователя с помощью imshow.
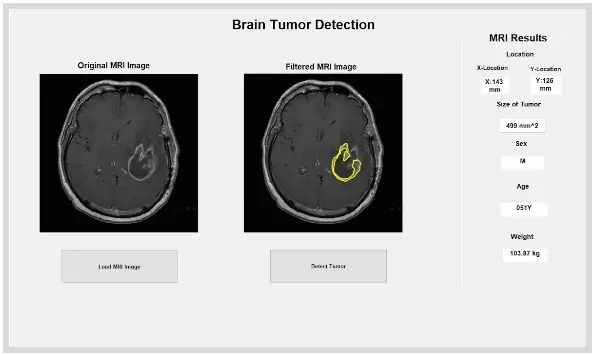
Встроенная функция MATLAB «dicominfo» также чрезвычайно полезна при обращении ко всей информации каждого файла dicom MRI. Мы использовали эту функцию для извлечения всей описательной информации о пациентах, такой как их пол, возраст, вес и рост. Эта функция также предоставляет вам порядок стека, который полезен для реализации программы в графическом пользовательском интерфейсе. Мы создали переменные для каждой описательной информации о пациентах, которые будут использоваться для графического интерфейса пользователя при нажатии кнопки обнаружения.
Шаг 3. Фильтрация изображений

После загрузки и чтения файла DICOM необработанного изображения изображение необходимо преобразовать из шкалы серого в бинаризованную форму, состоящую только из черных и белых пикселей. Мы использовали функцию imbinarize для создания двоичного изображения из необработанного изображения с помощью управление аспектами адаптивной пороговой обработки при значении чувствительности 0,59. Коэффициент пороговой чувствительности по умолчанию 0,5 был низким и не мог обнаружить более яркие капли и пятна на изображении, поэтому мы увеличили его до 0,59.
Затем преобразованное в двоичное изображение изображение обрабатывается через медианный фильтр с использованием функции «medfilt2», поскольку преобразованное в двоичное изображение изображение является двумерным. Мы устанавливаем каждый выходной пиксель так, чтобы он содержал среднее значение в окрестности 5 x 5 вокруг соответствующего пикселя во входном бинаризованном изображении. Это уменьшает шум и сохраняет края квадрата 5 x 5 вокруг каждого пикселя. Затем мы применяем скользящее окно с помощью «strel», чтобы создать плоский элемент структуры в форме диска с радиусом окрестности 2, чтобы идентифицировать каждый центральный, исходный пиксель в каждой окрестности диска. Мы использовали элемент структурирования диска, потому что мы анализируем каждое круглое пятно и пиксели в каждом пятне, поэтому элемент формы диска более полезен.
После того, как изображение было отфильтровано, его можно очистить с помощью функции «imclose», чтобы удалить черные пятна между отфильтрованными белыми пикселями в изображении и закрыть все зазоры вокруг него. Затем полностью обработанное изображение может быть нанесено на второй участок заранее выделенного рисунка, что позволяет сравнивать необработанное и отфильтрованное изображение.
Шаг 4: изоляция опухоли с помощью эллиптической маски


Яркие пятна опухоли затем можно выделить из основного отфильтрованного изображения с помощью заранее созданной эллиптической маски. Чтобы создать эту маску, вы должны знать размер исходного необработанного изображения сканирования МРТ и, используя длину строки и столбца в качестве координат x и y соответственно, назначить координаты центра эллиптического тренажера. Мы устанавливаем ось Y как большую ось с радиусом 50 единиц от центра и малую ось с радиусом 40 единиц от центра.
Мы использовали функцию MATLAB 'meshgrid' для создания декартовой плоскости с координатами двумерной сетки на основе координат, содержащихся в векторах от 1 до длины оси x и от 1 до длины оси y изображения.. Col - это матрица, в которой каждая строка является копией оси x, а Row - это матрица, в которой каждый столбец является копией оси y. Декартова сетка, представленная координатами Col и Row, имеет строки длины (1: Y_Size) и столбцов длины (1: X_Size). Используйте индексы Col и Row, сгенерированные декартовой сеткой, чтобы определить уравнение эллипса в зависимости от заданного радиуса и координат центра. Эллиптический контур теперь можно заполнить белыми пикселями, обнаруженными в пятнах опухоли.
Используя предварительно сгенерированную эллиптическую маску, мы можем вырезать конкретную опухоль, которую вы хотите проанализировать, из отфильтрованного изображения. Эллиптическая маска определяет, какие пятна логически вписываются в контур эллипса, и принимает это как пятно на отфильтрованном изображении как приемлемое в качестве опухоли. Затем функция «bwareafilt» отфильтровывает все другие объекты за пределами этой обнаруженной опухоли из изображения. Мы использовали конкретное окно 500 на 4000 эмпирически на основе размеров всех изображений. Затем мы применили другое скользящее окно со «стрелой» в качестве структурирующего элемента в форме плоского диска с большим радиусом окружения 6, чтобы закрыть зазоры между каждым центральным белым пикселем в обнаруженной опухоли. Обнаруженное пятно опухоли дополнительно очищается с помощью «imclose» для дальнейшего удаления черных пикселей и заполнения всех отверстий «imfill». Эта обработанная опухоль затем может быть отображена на третьем подзаголовке предварительно назначенного графика, чтобы обеспечить сравнение между изолированной опухолью и исходными и отфильтрованными изображениями МРТ-сканирования.
Шаг 5: очертание опухоли
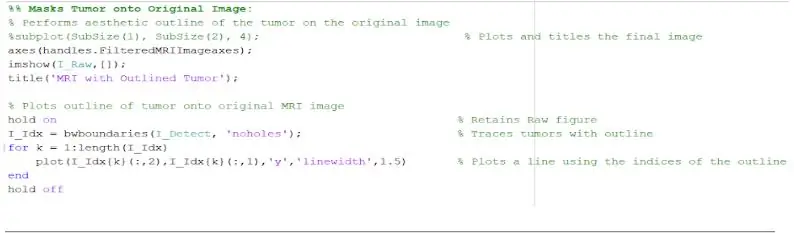
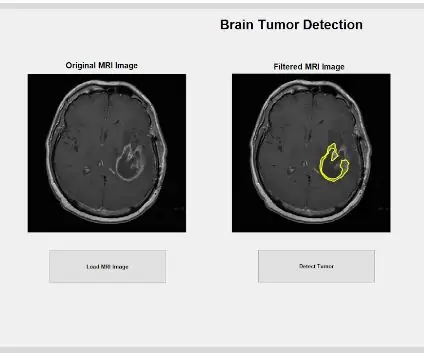
Теперь, когда опухоль изолирована с помощью маски, ее можно очертить и отобразить на исходном изображении, чтобы показать ее точное местоположение. Для этого мы использовали функцию «bwboundaries», чтобы проследить ранее обнаруженную опухоль по контуру. Мы указали, что контур не включает дыры внутри опухолевого объекта, когда он очерчивается. Его можно нанести на исходное необработанное изображение, используя цикл «for», который рисует контур вокруг опухоли с использованием индексов линии с шириной линии 1,5 пикселя. Затем этот контур наносится на необработанное изображение, показывая точный размер и расположение опухоли по сравнению с исходным МРТ-сканированием.
Шаг 6: Анализ физических свойств опухолей
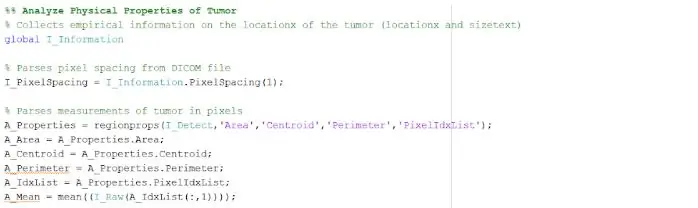
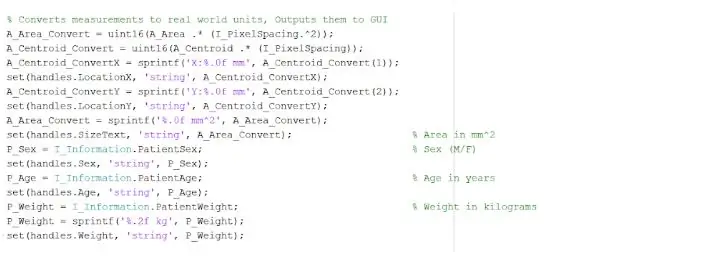
Изолированное и очерченное пятно может предоставить нам полезную информацию о размере, площади и местонахождении опухоли. Мы использовали функцию «regionprops», чтобы определить свойства опухоли, относящиеся к площади, периметру, центроидам и значению индекса пикселей. Это значение индекса пикселя дает нам реальные единицы измерения для каждого пикселя каждого изображения, уникальные для каждого сканирования. Эти свойства затем могут быть преобразованы в реальные единицы миллиметра. Эмпирическая информация, которую предоставляет нам программа, уникальна для каждого МРТ-сканирования и чрезвычайно полезна для определения размера, местоположения и типа опухоли, которую пользователи могут анализировать и включать в графический интерфейс.
Рекомендуемые:
USB-мод Honda Civic 10-го поколения 2018 для головного устройства Kenwood: 5 шагов

USB-мод Honda Civic 10-го поколения для головного устройства Kenwood 2018: в этой статье я модифицировал отверстие USB-порта моего Civic, чтобы принять тот, который я купил у Amazon, чтобы я мог подключить его к своему штатному головному устройству Kenwood (DMX9706S). Он находится в той же дыре, и его выполнение может занять 30 минут или больше
Обнаружение вибраций с помощью модуля пьезоэлектрического датчика удара: 6 шагов

Обнаружение вибраций с помощью модуля пьезоэлектрического датчика удара: в этом руководстве мы узнаем, как обнаруживать вибрацию удара с помощью простого пьезоэлектрического датчика Модуль вибрации и Visuino. Посмотрите демонстрационное видео
Обнаружение объектов с помощью Sipeed MaiX Board (Kendryte K210): 6 шагов

Обнаружение объектов с помощью досок Sipeed MaiX (Kendryte K210): в продолжение моей предыдущей статьи о распознавании изображений с помощью досок Sipeed MaiX Boards я решил написать еще один учебник, посвященный обнаружению объектов. Недавно появилось интересное оборудование с чипом Kendryte K210, в том числе S
Вход DIY Aux для штатного головного устройства автомобиля: 5 шагов (с изображениями)

DIY Aux Input для штатного автомобильного головного устройства: если вы хотите играть на своем телефоне или ipod (они все еще делают их) и других аудиоплеерах, а ваше головное устройство - это старый AF, то это решение, которое вы ищете
Ard-e: робот с Arduino в качестве мозга: 9 шагов

Ard-e: робот с Arduino в качестве мозга: как создать робота с открытым исходным кодом, управляемого Arduino, менее чем за 100 долларов. Надеюсь, после прочтения этого руководства вы сможете сделать свой первый шаг в робототехнике. Ard-e стоит от 90 до 130 долларов в зависимости от того, сколько у вас запасной электроники
