Оглавление:
- Шаг 1. Использование Brain Box
- Шаг 2: Введение: анализ поперечных сечений
- Шаг 3: Настройка функции: LevelCurveTracings.m
- Шаг 4: Разработка алгоритма привязки: поиск границ
- Шаг 5: Разработка алгоритма привязки: создание массива внешних границ
- Шаг 6: Разработка алгоритма привязки: работа с центральной точкой
- Шаг 7: Разработка алгоритма привязки: когда центроид не отцентрирован
- Шаг 8: Разработка алгоритма привязки: пересечение отверстий
- Шаг 9: Разработка алгоритма привязки: определение местоположения отверстий, завершение границ мозга и границ отверстий
- Шаг 10: Регистрация данных: функция PatientFiles.m
- Шаг 11: Запись данных в файлы
- Шаг 12: Регистрация данных: отображение графика объема мозга с течением времени
- Шаг 13: Устранение пробелов в подзаголовках: Subplotclose.m
- Шаг 14: Основной код: очистка всего и запрос ввода
- Шаг 15: Основной код: пакетная обработка изображений
- Шаг 16: Основной код: заполнение
- Шаг 17: Основной код: определение границ
- Шаг 18: Основной код: определение правильного размера Z
- Шаг 19: Основной код: определение координат X и Y
- Шаг 20: Основной код: построение трехмерной структуры, поиск объема и запись данных
- Автор John Day day@howwhatproduce.com.
- Public 2024-01-31 10:24.
- Последнее изменение 2025-01-23 15:05.
Продвижение к рубежу более продолжительной человеческой жизни вызвало рост болезней, невиданных цивилизациям до нашей. Среди них от болезни Альцгеймера в 2017 г. пострадало примерно 5,3 миллиона живущих пожилых американцев, или примерно 1 из 10 пожилых американцев (https://www.alz.org/facts/), а также бесчисленное количество других людей, страдающих деменцией. Чтобы помочь в борьбе за понимание того, что беспокоит наших старших, этот код предоставит будущим исследователям и любопытствующим возможность отслеживать объем мозга с течением времени.
Шаг 1. Использование Brain Box
Чтобы использовать мозговой ящик, нужно только следующее:
- МРТ головного мозга, а также имя и формат таких файлов (все они должны иметь примерно одинаковые размеры).
- Длина одного сканирования
- Расстояние между слоями (МРТ)
- Имя пациента (при вводе не включайте пробелы и используйте имя и фамилию с заглавной буквы, например: ИмяФамилия)
Таким образом, у человека появляется возможность отслеживать тенденции изменения объема мозга у отдельных людей с течением времени. Таким образом, это программное обеспечение позволяет отслеживать тенденции развития болезни Альцгеймера. Длина, которую мы использовали в испытании, составляла 180 мм для длины одного сканирования и 5 мм для расстояния между сканированиями МРТ, исходя из средних значений.
Однако применение мозговой коробки не должно ограничиваться этой единственной задачей. Если поперечные сечения данного твердого тела являются фотографиями, как и сама опухоль, тенденции изменения объема для них также можно отслеживать с помощью программного обеспечения.
Шаг 2: Введение: анализ поперечных сечений
В трехмерных структурах двумерные плоскости, из которых они состоят, будут называться поперечными сечениями. Представьте себе, что стопка бумаг составляет прямоугольную призму, тогда каждый лист бумаги будет поперечным сечением бумаги. Представляя мозг, мы применяем тот же образ мышления. МРТ (магнитно-резонансная томография) (см. Информацию о МРТ) захватывает поперечные сечения мозга, и, используя границы, определенные в каждом «слое» мозга, мы можем построить структуру для моделирования и определения объема мозга.. Однако сначала мы должны создать функцию для предоставления информации о таких границах.
Шаг 3: Настройка функции: LevelCurveTracings.m
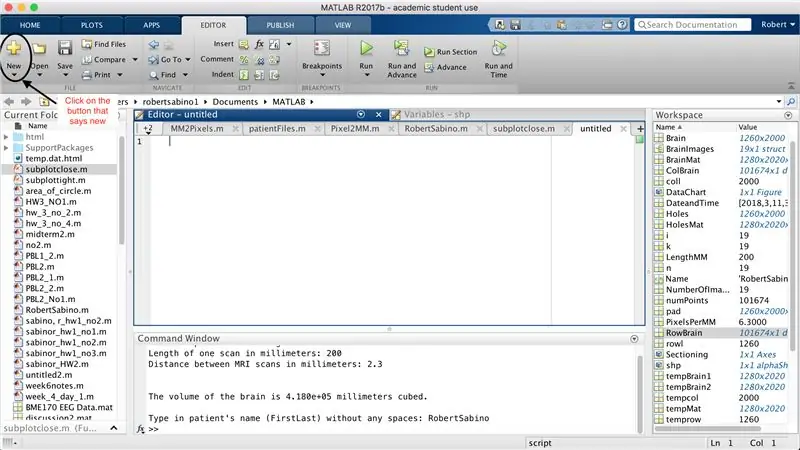
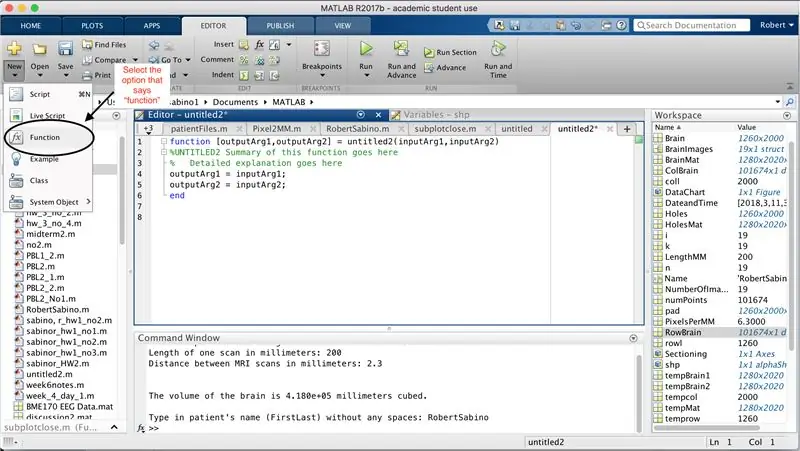
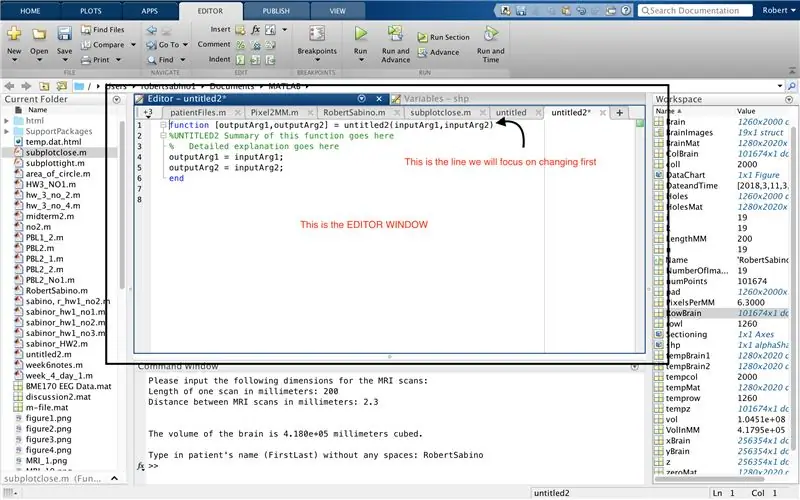

Во-первых, убедитесь, что на ваш компьютер загружен MATLAB_R2017b (загрузите здесь) и откройте MATLAB. В интерфейсе MATLAB нажмите кнопку в верхнем левом углу окна с надписью «New» с полужирным желтым знаком плюс и выберите параметр «функция», чтобы открыть в окне редактора пространство, напоминающее пространство в третья картинка. Мы сосредоточимся на изменении первой строки для настройки функции. Там, где написано "outputArg1", замените его на "brain", "outputArg2", чтобы сказать "дыры", "untitled2" на "exp2" и "inputArg1" на "image", и удалите "inputArg2". Теперь у вас есть функция, которую нужно вызвать с помощью «exp2», принимая один аргумент «image» и выводя границы «мозга» и «дыр». Первая строка функции должна напоминать линию, изображенную на четвертом рисунке. Удалите весь код под этой начальной строкой.
Шаг 4: Разработка алгоритма привязки: поиск границ

Введите следующий код под строкой. В этом разделе функции построчно выполняется следующее.
- Загрузите изображение «image» в переменную «mri».
- Превратите «мрт» в изображение, состоящее из значений в диапазоне от единиц до единиц и нулей (также называемое бинаризацией) на основе установленного порогового значения. Если значение в пикселе равно или больше 0,1, оно устанавливается в единицу, если нет, значение в этом пикселе устанавливается в ноль.
- Следующие четыре строки превращают 10 столбцов и строк по краям слоя сканирования МРТ в нули, чтобы избежать считывания неправильных значений как ограничивающих (как было показано в ходе экспериментов с кодом).
- В последней строке bwboundaries отслеживает границы бинаризованного изображения «mri» и устанавливает его равным «b», массиву с элементами, чьи индексы соответствуют тем из границ, установленных на единицу.
Шаг 5: Разработка алгоритма привязки: создание массива внешних границ
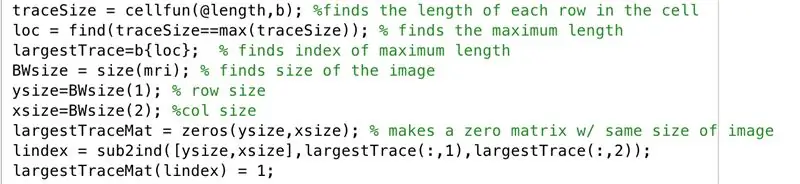
Следуйте инструкциям в окне редактора, используя следующий код на картинке. В этом разделе кода построчно выполняется следующее.
- Найдите длину каждой из строк бинаризованного изображения «b» (cellfun применяет длину функции к каждой строке).
- Установите "loc", чтобы сохранить максимальную длину.
- Найдите индекс максимальной длины, установленной для хранения в "largeTrace".
- Найдите размер изображения «mri», которое имеет тот же размер, что и «b», и установите значение «BWsize».
- Найдите количество строк в массиве изображения, задав значение «ysize».
- Найдите количество столбцов в массиве изображения, задав значение «xsize».
- Сгенерируйте массив «largeTraceMat», матрицу нулей «ysize» на «xsize».
- Найдите эквивалентный индекс из значений с индексами, соответствующих наибольшим значениям x и y, сохраните в векторе "lindex".
- В матрице нулей «largeTraceMat» превратите элементы с индексами, которые соответствуют значениям индекса, хранящимся как элементы в «lindex», в единицы.
Таким образом, логический массив «наибольшийTraceMat» имеет наибольшую ограниченную область данного поперечного сечения сканирования мозга, отображенную в виде единиц на фоне нулей.
Шаг 6: Разработка алгоритма привязки: работа с центральной точкой

Затем мы должны проверить, состоит ли поперечное сечение более чем из одной области (самой большой). Проверяя выравнивание центроида самой большой области, мы можем увидеть, есть ли одна смежная область, которая даст более центрированный центроид, или возможность нескольких областей.
- Используйте "regionProps", чтобы найти информацию о присутствующих центроидах, установите равным массиву структур "tempStruct"
- Сформировать массив "centroids" с данными из поля "centroid", соединенными по вертикали.
- Возьмите второй столбец значений «центроидов» (координаты горизонтального измерения).
- Запустите фильтр, чтобы проверить выравнивание центроида относительно горизонтального центра.
Шаг 7: Разработка алгоритма привязки: когда центроид не отцентрирован
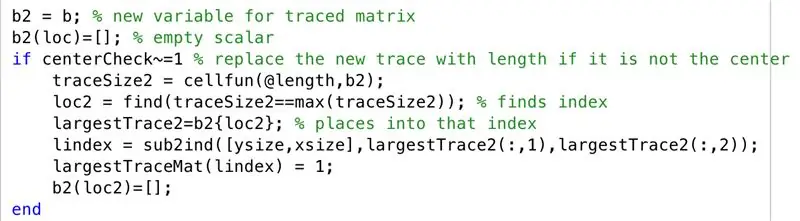
В сценарии, когда центр тяжести самой большой области трассы не центрирован, мы выполняем следующие шаги. Как мы наблюдали при сканировании МРТ, тенденция заключалась в том, чтобы полушария мозга отображались в поперечном сечении, когда они не были смежными, поэтому теперь мы продолжаем наносить второй по величине след вместе с самым большим следом в «largeTraceMat».
- Установите для отслеживаемой матрицы новую переменную "b2"
- Инициализировать пустую матрицу "b2" набором, индексированным "loc"
- Создайте условие, когда центроид не отцентрован (например, слой с несколькими регионами)
- Установите новый размер трассировки для каждой строки (traceSize2)
- Установите "loc2", чтобы найти индексы, в которых есть границы.
- Пусть ячейки, указанные в "loc2" в "b2", равны "largeTrace2"
- Преобразуйте индексы в индексы, установите "lindex"
- Измените элементы, соответствующие "lindex" в "largeTraceMat", на 1
- Инициализировать пустую матрицу "b2" набором, индексированным "loc2"
Шаг 8: Разработка алгоритма привязки: пересечение отверстий

При работе с дырами значения, хранящиеся в «b2», отслеживали структуры, отличные от самой большой трассы, и нанесение их на заполненную форму «mostTraceMat» выявит, где есть дыры в областях мозга.
- Создайте массив "fillMat", который представляет собой заполненную форму "largeTraceMat".
- Создайте массив "InterferenceMat", массив нулей "ysize" на "xsize".
- Создайте массив "Interferenceloc" для хранения значений из "b2", соединенных по вертикали.
- Создайте массив «lindex» для хранения индексов, соответствующих «Interferenceloc».
- Для индексов в "InterferenceMat", которые соответствуют "lindex", установите значение 1, чтобы создать другую ограниченную область.
Шаг 9: Разработка алгоритма привязки: определение местоположения отверстий, завершение границ мозга и границ отверстий

- Установите для массива «tempMat» значение «InterferenceMat» плюс «fillMat», таким образом добавляя каждое значение в матрице друг к другу.
- Установите массив "HoleLoc" равным индексам, где "InterferenceMat" и "fillMat" были равны единице.
- Установите "HoleMat" как нулевую матрицу размеров "ysize" x "xsize"
- Установить индексы в "holeMat", которые равны "holeLoc", как единицы
- Установите для "мозга" значение "largeTraceMat".
- Установите "Hole" на "HoleMat".
При обнаружении, когда значения добавленных матриц были равны 2, расположение отверстий было легко зафиксировано и нанесено на пустую матрицу.
Шаг 10: Регистрация данных: функция PatientFiles.m
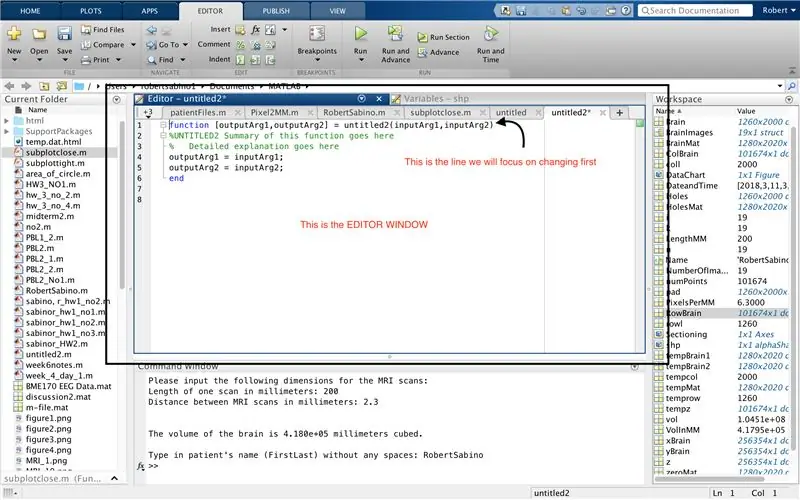

Подобно настройке последней функции, нажмите кнопку в верхнем левом углу окна с надписью «New» с жирным желтым знаком плюса и выберите параметр «функция», чтобы открыть в окне редактора пространство, которое напоминает то, что на третьей картинке. В первой строке удалите матрицу вывода и замените просто «output», замените «untitled2» на «PatientFiles», удалите все входные аргументы и вместо этого выполните форматирование, указанное на четвертом изображении строки кода. Первая строка этой функции должна соответствовать форматированию изображения.
Шаг 11: Запись данных в файлы
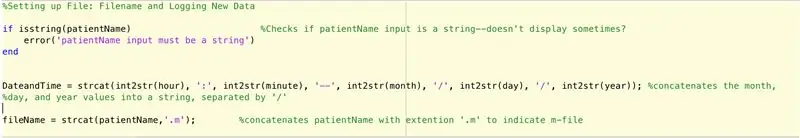

Чтобы настроить файл для регистрации данных, найденных основной функцией (еще не описанных), мы должны выполнить следующие шаги (как предписано в коде построчно).
- Проверьте, является ли ввод для имя пациента строкой.
- Если это не строка, отобразите, что вводимя пациента должна быть строка.
- Завершите оператор if (предотвращение ошибки).
- Установите строковое выражение «DateandTime», которое будет иметь следующий формат: час: минуты - месяц / день / год.
- Задайте для переменной fileName следующее: имя пациента.m.
Теперь перейдем к следующему разделу функции: существует ли уже файл с таким именем?
1) Предположим, файл с таким именем уже существует:
- Запустите файл, чтобы получить значения из прошлого в очереди.
- Добавьте данные «DateandTime» текущей итерации как новую ячейку в массив ячеек значений x (конец индекса + 1)
- Добавьте текущее значение brainVolume как новую ячейку в массив ячеек значений y (конец индекса + 1)
- Сохраните текущие переменные, загруженные в файл.
2) Предположим, файла с таким именем не существует:
- Создайте новый файл с именем, хранящимся в переменной «имя пациента».
- Добавьте текущие данные DateandTime в виде ячейки в пустой массив ячеек со значениями x.
- Добавьте текущие данные "brainVolume" как ячейку в пустой массив ячеек значений y.
- Сохраните текущие переменные, загруженные в файл.
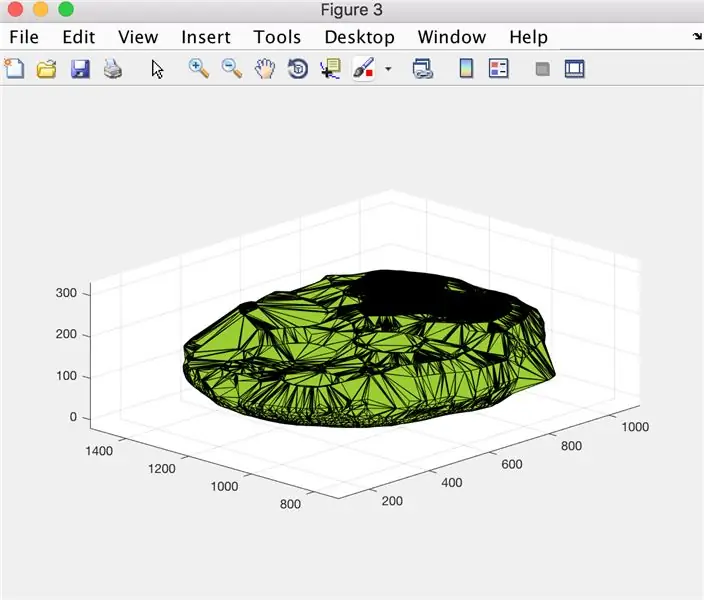
Шаг 12: Регистрация данных: отображение графика объема мозга с течением времени

- Преобразуйте массив значений x (xVals) в категориальный массив (xValsCategorical), чтобы можно было построить график.
- Окно создания рисунка 5
- Постройте точки, обозначенные «xValsCategorical» и «yVals» (содержащие объем мозга), используя полые кружки для обозначения точек и соединенных пунктирными линиями.
- Назовите график как: имя пациента, данные об объеме мозга.
- Обозначьте ось x, как показано на рисунке.
- Обозначьте ось Y, как показано на рисунке.
- Пусть цифра 5 равна output
Отсюда вызываемая функция PatientName даст файл с отредактированными данными, отслеживающими объем мозга с течением времени, и график, отображающий тенденции.
Шаг 13: Устранение пробелов в подзаголовках: Subplotclose.m

Функция, адаптированная из кода с https://www.briandalessandro.com, работает, чтобы закрыть пробелы между фигурами подзаголовка основного кода, когда создаются фигуры, отображающие изображения МРТ и слои мозга. Функция subplot, используемая в subplotclose.m, регулирует положение данных subplot, чтобы они плотно прилегали друг к другу в аспекте более длинного измерения. Например, если код предполагает матрицу 7 x 3, строки будут располагаться плотно, поскольку размер строки больше. Если код предполагает матрицу 3 x 7, столбцы будут плотно прилегать друг к другу с зазорами в строках, как показано на рисунках нашего основного кода.
Шаг 14: Основной код: очистка всего и запрос ввода

Чтобы запустить основной код, нажмите ту же кнопку с надписью «New» в верхнем левом углу окна и выберите «Script» вместо «Function» из предыдущих разделов. Введите код, как показано на рисунке, в окне редактора. Строки кода выполняют следующие задачи по порядку:
- Закройте все открытые файлы, кроме файлов 0, 1 и 2.
- Закройте все окна с фигурами.
- Очистите все переменные в рабочей области.
- Очистите командное окно.
- Отображение в командном окне: введите следующие размеры для МРТ-сканирования:
- В новой строке в Командном окне спросите: Длина одного сканирования в миллиметрах:. Ответ, введенный пользователем, будет установлен на переменную lengthMM.
- В новой строке спросите: Расстояние между снимками МРТ в миллиметрах:. Ответ, введенный пользователем, будет установлен в переменную «ZStacks».
Шаг 15: Основной код: пакетная обработка изображений

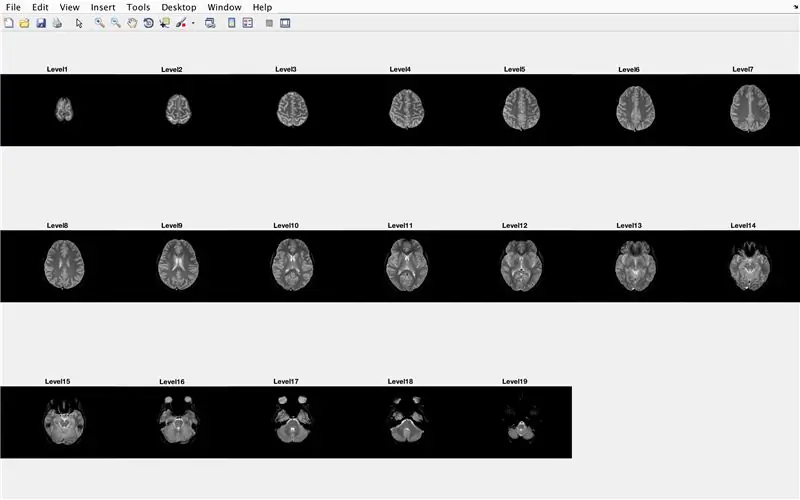
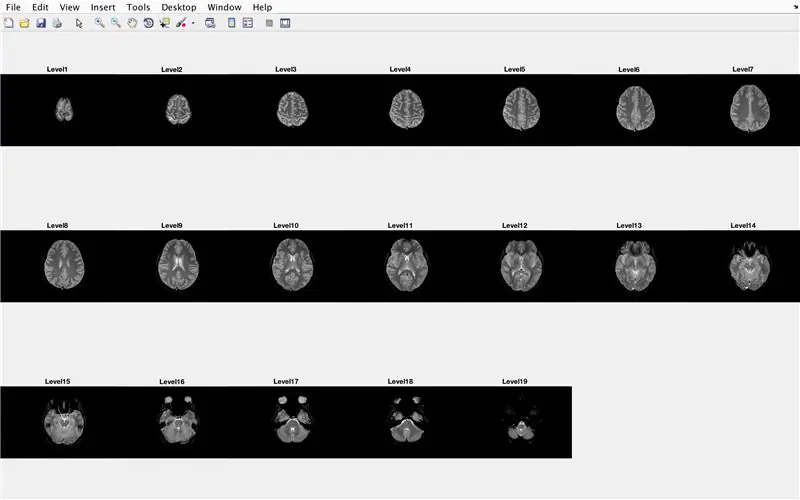
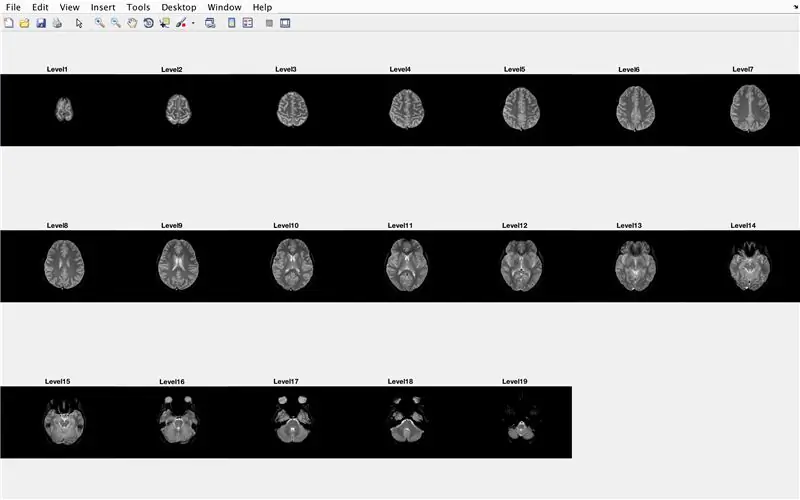
В этом разделе код загрузит изображения (состоящие из МРТ-сканирований поперечных срезов головного мозга) и сохранит имена каждого файла изображения в переменной «Base» и отобразит каждое из МРТ-сканирований. Пожалуйста, введите код на картинке, который выполняет следующие действия:
- Создайте структурный массив BrainImages, содержащий информацию обо всех файлах в текущей папке, которые соответствуют формату имени MRI _ (). Png
- Установите переменную NumberofImages равной количеству элементов в массиве структур BrainImages.
- Открыть окно рисунка 1
- Установите цикл for для циклического перебора количества изображений, подсчитанных в файле.
- Для каждого цикла CurrentImage - это соответствующее имя каждого файла MRI_i-p.webp" />
- Создайте подзаголовок 3 x 7 для отображения 19 изображений, которые будут загружены с помощью "imshow".
- Отображать каждое изображение как другой элемент в окне рисунка подзаголовка
- Назовите каждый элемент подзаголовка как Level_, где blank - номер итерации цикла for.
- Завершите цикл for (избегая ошибки)
Это отобразит в окне рисунка 1 все снимки МРТ в необработанном виде в конфигурации 3 x 7 без зазоров в ориентации x.
Шаг 16: Основной код: заполнение

Используя заполнение, мы избегаем проблемы небольших расхождений в размерах изображений, которые могут привести к ошибке несоответствия размеров в случае, если одно изображение немного больше другого.
- Открыть окно рисунка 2
- Загрузите матрицу изображения из MRI_1-p.webp" />
- Найдите размер матрицы изображения и установите «OriginalXPixels» (для количества строк) и «OriginalYPixels» (для количества столбцов).
- Настройте матрицу «BrainMat» так, чтобы она состояла из всех нулей, с дополнительными 20 строками и еще 20 столбцами для каждой плоскости и 19 полными сечениями, по одному на каждую плоскость.
- Настройте «HolesMat», чтобы он состоял из того же трехмерного массива нулей, чтобы ввести координаты отверстия позже.
- Создайте "zeroMat", равный размеру прокладки плюс двадцать строк и двадцать столбцов, двумерный массив нулей.
Шаг 17: Основной код: определение границ
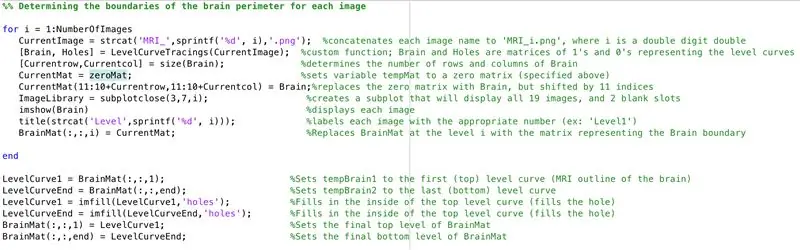
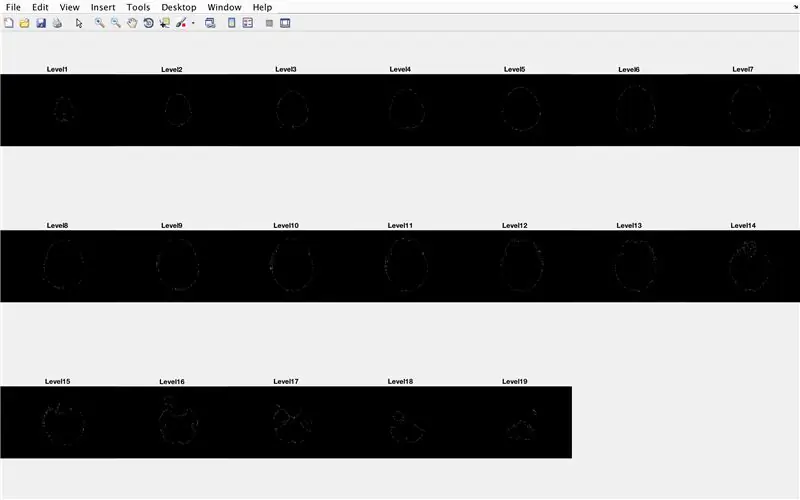
- Установите цикл for для просмотра данных из каждого изображения, загруженного ранее
- Таким же образом, как и ранее при пакетной обработке, «CurrentImage» загружает файлы с «MRI_i.png», где i - номер итерации.
- Пропустите каждое изображение через функцию обработки "LevelCurveTracings2.m", которую вы создали ранее.
- Найдите размер вывода «Мозг», установите количество строк на «Currentrow» и количество столбцов на «Currentcolumns».
- Установите "CurrentMat" на матрицу нулей с размерами, указанными в "Currentrow" и "Currentcolumns".
- Центрируйте данные из "Brain" в "CurrentMat" с полем в 10 строк со всех сторон.
- Создайте подзаголовок размером 3 x 7, чтобы отобразить границы изображений.
- Назовите каждый из элементов подзаголовка в окне рисунка
- Сгенерируйте трехмерную матрицу «BrainMat», состоящую из каждого граничного слоя «CurrentMat».
- Завершите цикл for (чтобы избежать ошибок)
В следующем подразделе заполняются отверстия слева вверху и внизу предлагаемой трехмерной формы.
- Установите «LevelCurve1» равным первому слою «BrainMat» (нижняя часть твердого тела).
- Установите «LevelCurveEnd» равным последнему слою «BrainMat» (верх твердого тела).
- Заменить "LevelCurve1" заполненным слоем
- Заменить "LevelCurveEnd" заполненным слоем
- Установите слой с заливкой как нижний слой "BrainMat".
- Установите слой с заливкой как верхний слой "BrainMat".
Шаг 18: Основной код: определение правильного размера Z

Первые три строки состоят из настройки пустого массива «z» и выполнения простых операций преобразования (деление пикселей по длине) для получения правильного значения объема в мм ^ 3.
- Создайте цикл for для циклического просмотра каждого слоя
- Найдите количество единиц в данном слое
- Преобразуйте координаты z для единиц в значения, которые масштабируются до надлежащего соотношения, установленного на "tempz", вектор-столбец
- Добавьте значение z для кривой уровня к вектору z
При этом координаты z корректируются правильно.
Шаг 19: Основной код: определение координат X и Y

Теперь определим положения x и y каждой из точек границ.
- Инициализировать "xBrain" как пустой массив
- Инициализировать yBrain как пустой массив
- Настройте цикл for для просмотра каждого загруженного изображения
- Скомпилируйте матрицу из двух столбцов для хранения плоских координат каждой точки границы, представленных векторами-столбцами «RowBrain» и «ColumnBrain».
- Добавьте "xBrain" к найденным в данный момент координатам "RowBrain".
- Добавьте yBrain к найденным в настоящий момент координатам ColumnBrain.
- Завершите цикл for (чтобы избежать ошибки)
Шаг 20: Основной код: построение трехмерной структуры, поиск объема и запись данных
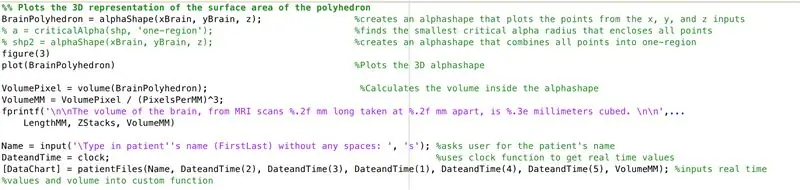

Используя функцию alphaShape, мы создадим трехмерную структуру, по которой мы сможем вычислить объем мозга.
- Используйте функцию alphaShape, вставьте векторы «xBrain», «yBrain» и «z» в качестве координат x, y и z и установите равным «BrainPolyhedron»
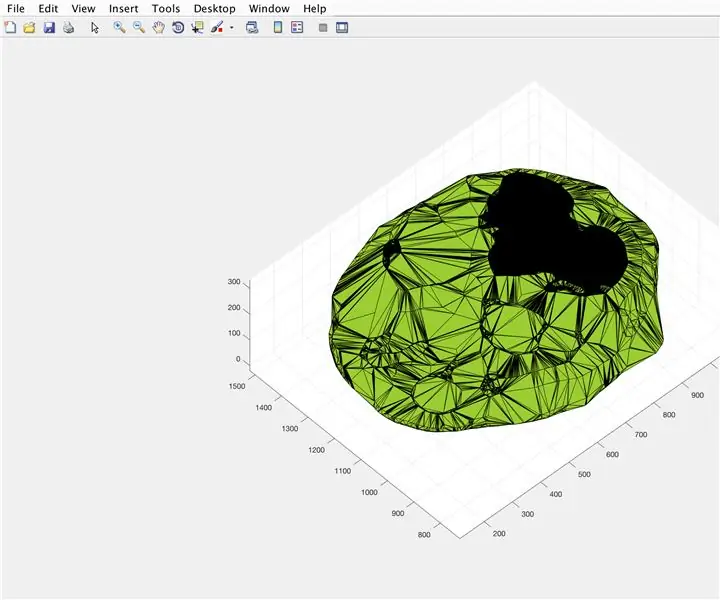
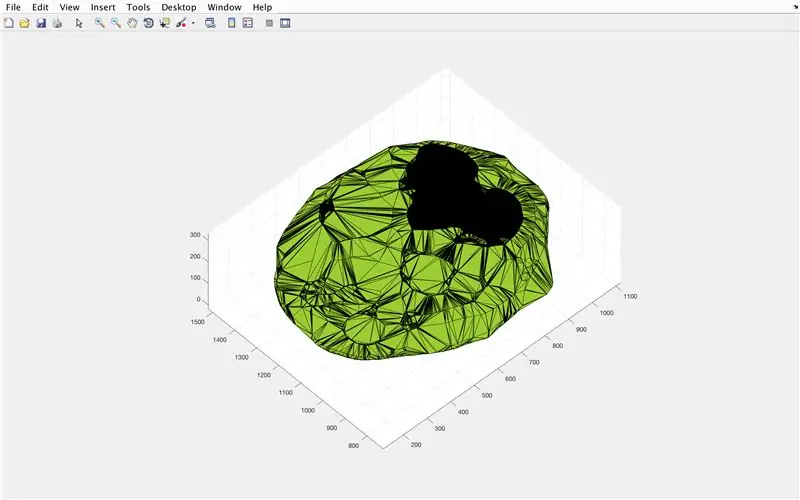
- Открыть окно рисунка 3
- Постройте вычисленную альфа-форму «BrainPolyhedron», отобразите в окне рисунка
- Рассчитайте объем альфа-формы, используя функцию «объем», которая работает для альфа-фигур.
- Преобразовать объем в мм ^ 3
- Распечатайте объем твердого тела в Командном окне
- Запрос на ввод имени пациента
- Получите текущую дату и время с часами и установите "DateandTime"
- Вызовите функцию «PatientFiles», чтобы записать и отобразить рассчитанные данные.
Отсюда второе и третье изображения должны показывать всплывающие фигуры, а четвертое изображение должно отображаться в Командном окне.
Рекомендуемые:
Как сделать отслеживание данных о коронавирусе COVID 19 в реальном времени с помощью ESP8266, отображение электронной бумаги: 7 шагов

Как сделать трекер данных о коронавирусе COVID 19 в реальном времени с ESP8266, дисплей электронной бумаги: 1
Создайте считыватель объема резервуара менее чем за 30 долларов с помощью ESP32: 5 шагов (с изображениями)
Создайте счетчик объема резервуара менее чем за 30 долларов с помощью ESP32: Интернет вещей принес множество ранее сложных приложений для устройств в дома многих пивоваров и виноделов. Приложения с датчиками уровня десятилетиями использовались на крупных нефтеперерабатывающих заводах, водоочистных установках и в химической
Настройка часов реального времени DS3231 RTC (часов реального времени) точно, быстро и автоматически с помощью Java (+ -1 с): 3 шага

Установка часов реального времени DS3231 RTC (часов реального времени) точно, быстро и автоматически с использованием Java (+ -1 с): это руководство покажет вам, как установить время на часах реального времени DS3231 с помощью Arduino и небольшого приложения Java, которое использует последовательное соединение Arduino. Основная логика этой программы: 1. Arduino отправляет последовательный запрос
Куб времени - гаджет для отслеживания времени Arduino: 6 шагов

Time Cube - гаджет для отслеживания времени Arduino: я хотел бы предложить вам простой, но действительно полезный проект Arduino для отслеживания событий времени, перевернув какой-нибудь интеллектуальный гаджет-куб. Переверните его на "Работа". > " Учиться " > " Работа по дому " > " Отдых " сторона, и он посчитает
Отслеживание GPS в реальном времени: 3 шага
Отслеживание GPS в реальном времени: в этом руководстве мы покажем вам, как отслеживать местоположение в реальном времени с помощью BerryGPS-GSM, Raspberry Pi Zero и Initial State. Мы отправим долготу, широту и скорость через 3G с помощью BerryGPS-GSM в исходное состояние
